Epigenetics Sequencing |
染色质免疫共沉淀技术(Chromatin Immunoprecipitation),是一类通过将与特定蛋白质与其结合DNA序列共同沉淀下来研究其相互作用的 方法,这一方法和测序技术结合起来,可以研究转录因子在基因组上的结合区域、特异性修饰的组蛋白位点以及其他DNA结合蛋白的基因组 特异结合位点等表观遗传性学问题。ChIP-Seq技术是在ChIP-on-Chip的基础上发展来的,相对于ChIP-on-Chip来说,ChIP-Seq有很多优 势:更高的通量;更低廉的价格;双通道ChIP-Seq可以弥补单通道带来的GC偏向问题,产生更加可靠的数据。
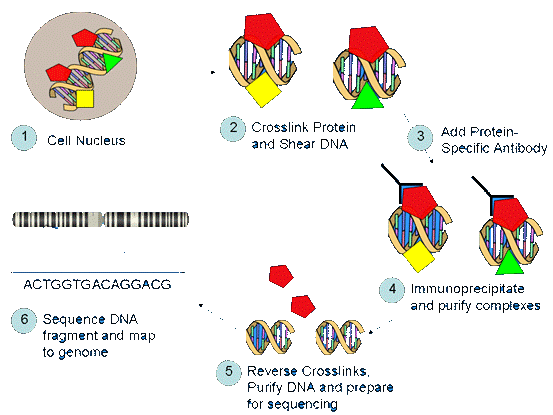
数据分析项目
一.数据基本质量分析
基于不同的技术平台,所需要的分析方法不尽相同。对于一般的单通道测序,我们进行序列长度统计、丰度统计、定位百分比(mapping coverage)统计等等,通过随机mapping,我们可以对数据质量进行估计。对于有Input lane的双通道测序,我们会进行横向的对比以得到更准确的结果。
二.峰值的定位
对于不同的数据质量,可以选择相对合适的mapping方法,包括允许不同的mismatch数目等等以得到比较可靠的数据。然后基于这些定位到基因组上的序列和其丰度,找到那些有统计学意义的位点峰值(peak calling)
三.Binding motif分析
一般的DNA结合因子都是特异性的,有特定的结合基序,对于检测到的峰值序列,我们进行DNA binding motif分析,以期验证或者得到新的DNA结合因子binding motif。
四.和annotation library的分析对比
Peak calling在基因组中的位置在已知注释信息中的分布可以显示DNA结合蛋白的结合偏好性,我们会给出基于不同注释库的分布信息。
五.基因本体学分析(Gene Ontology Analysis)
对于特定的转录因子,其DNA结合位点的下游转录基因在特定试验条件下可能会行使相似的功能,GO分析会找出富集的那些可能的功能。
MeDIP(Methylated DNA Immunoprecipitation Sequencing )是通过使用5’-甲基胞嘧啶抗体富集高甲基化的DNA片段,该过程称为甲基化DNA 免疫共沉淀(MeDIP), 经MeDIP 富集的DNA 片段用Cy3 标记, 未被MeDIP富集的DNA 片段用Cy5 标记,接着把这些DNA片段直接测序,即MeDIP Sequencing。这种方法能同时检测整个基因组和特定位点的甲基化状态,从而比较不同细胞、组织、甚至疾病样本间的DNA甲基化修饰模式的差异。
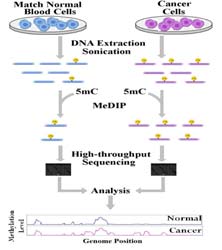
数据分析项目:
一.测序质量分析
对测序所得结果进行初步分析,包括序列reads数目、序列长度分布、可以定位到基因组的序列百分比、覆盖度、数据质量优劣等等重要信息。
二。 全基因组甲基化数据分布趋势分析
分析各条染色体中甲基化水平, 统计不同基因区域内的甲基化水平
三。 全基因组DNA 甲基化图谱
统计 染色体水平的甲基化C碱基的密度分布以及不同基因组区域的甲基化分布特征。
产品列表
共有 4 件 每页显示20件 第1页/共1页 1